很多从事核酸和蛋白等序列对比的专业人士需要进行本地序列对比,那么你可以下载Blast绿色版来实现哦,它是一款功能实用的本地序列对比软件,使用它可以让你在不接入互联网的环境下将比对的序列与本地数据库中的序列进行比对,主要是用作核酸和蛋白等序列对比,下面还有本地化实现方法,大家可以按下面的操作流程进行哦,那么快来下载Blast免费版进行相关使用吧。
使用教程
Linux下BLAST+的本地化方法教程(NCBI-BLAST 2.2.29+单机运行操作流程)
本人在以下环境亲测有效:CentOS Linux 64 Bit
本文涉及到的路径需根据自己的电脑进行修改哦。
Linux下BLAST+的本地化(NCBI-BLAST 2.2.29+):
1. 下载软件BLAST:
在以下网址 ftp://ftp.ncbi.nlm.nih.gov/blast/executables/blast+/LATEST/
下载:
ncbi-blast-2.2.29+-x64-linux.tar.gz (根据自己的操作系统选择)。
2. 解压:
解压以后放在任意目录下都可以,把相应路径加入PATH变量就是。
例如解压到用户的主目录(/home/yonpen)下,将解压之后的文件夹重新命名为blast,则BLAST+的所有程序在目录/home/yonpen/blast/bin下。
3. 添加环境变量:
打开终端(Terminal),切换为root用户,执行vim /etc/profile (需了解Vim编辑器的基本命令)。
在最末尾添加:
export PATH=/home/yonpen/blast/bin:$PATH
保存退出。(环境变量的值由Blast所在路径决定。)
此处如果成功,注销之后执行blastn -version会出现版本信息(一定要先注销或者是重启电脑)。
4. 新建:
在目录/home/yonpen/blast下新建一个文件夹,命名为db 。
在/home/yonpen下新建一个文件,命名为.ncbirc。(文件名是以点号开头的)
在文件中添加内容:
[BLAST]
BLASTDB=/home/yonpen/blast/db
5. 下载FASTA格式的数据库:
ftp://ftp.ncbi.nlm.nih.gov/blast/db/FASTA/
比如下载nr.gz。
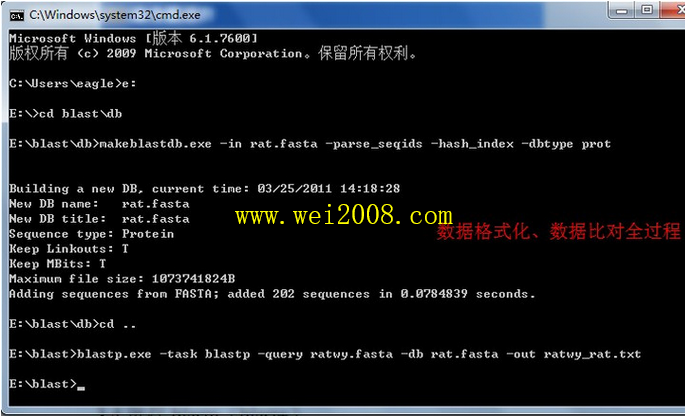
6. 建立BLAST+可用的数据库:
打开终端(Terminal),切换到/home/yonpen/blast/db目录下,执行(以蛋白质库nr为例):
makeblastdb –in nr -parse_seqids -hash_index -dbtype prot
(需自己输入,复制这行命令可能不行,不知道为何)
7. 使用程序:
如使用psiblast
在目录/home/yonpen/blast下新建3个文件夹,分别命名为pssm,input,output
设待查询序列所在文件的名字为a.fasta(一个文件放一条序列,并且必需为fasta格式)
执行命令:
psiblast -comp_based_stats 1 -evalue 0.001 -num_iterations 3 -db nr -query input/a.fasta -out output/a.txt -out_ascii_pssm pssm/a.pssm

 blast序列比对软件本地单机版2.2.30绿色版
blast序列比对软件本地单机版2.2.30绿色版